阿部班の機械学習を用いた染色体解析についての論文がScientific reports誌に掲載されました。
染色体を顕微鏡で観察することで行う「染色体解析」は染色体に起こる様々な異常 (染色体断裂、姉妹染色分体間接着異常、姉妹染色分体交換、染色体異数化など) を検出することができます。しかしながら正確に染色体解析を行うためには、その動物種の染色体の形状を正しく理解している必要があり、解析者の訓練には時間がかかります。また定量的な解析で統計的な有意差を出すためには非常に多くの染色体を解析しなければならず、それには膨大な時間と労力が必要となります。
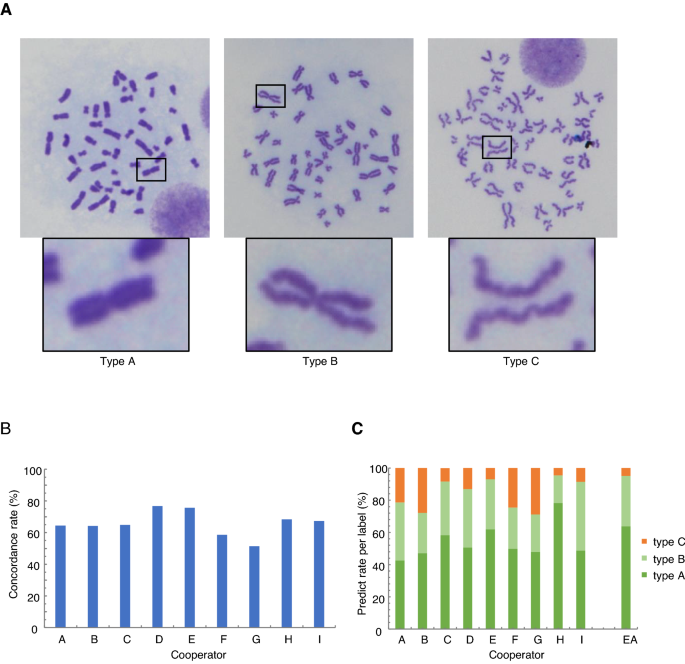
今回、阿部班員らは、畳み込みニューラルネットワークを用いた機械学習により、姉妹染色分体間接着に異常の見られる染色体を自動的に分類することに挑戦しました。SqueeezeNetという事前学習モデルに対し、600枚以上の染色体写真を使って学習させることで、最終的には人間が行った染色体解析との一致率を73.1%まで上昇させることができました。また、姉妹染色分体間接着において重要な役割を果たすことが報告されているCTF18を欠損させた細胞を作製し、本モデルでその染色体を解析させたところ、野生型細胞との間に違いを見出すことができました。現在、更なる分類精度の向上を目指してモデルの改良に取り組んでいます。
姉妹染色分体間接着の異常は染色体異数化の原因でもあり、Y染色体の喪失にもつながることから、本論文の成果が性染色体サイクル研究の発展に貢献することが期待されます。